TurboMole ist ein Programmpaket für ab-inito-Elektronenstrukturrechnungen. Aktuell auf unserem HPC System ist zur Zeit die Version 7.3.1.
- Offizielle Webseite: http://www.turbomole.com
- Manual: http://www.turbomole-gmbh.com/manuals/version_6_5/Documentation_html
TurboMole ist unter Umständen nicht ganz so intuitiv bedienbar, wie bestimmte amerikanische Alternativen. Da das HPC-Team aber einen quantenchemischen Hintergrund hat und selber TurboMole einsetzt, stehen wir gerne mit Rat und Tat zur Seite.
TurboMole kommt in drei Versionen daher:
- Sequential: Keine Parallelisierung, es wird immer nur 1 Core genutzt.
- Shared Memory / OpenMP: Parallelisierung über mehrere Cores, die einen gemeinsamen Speicherbereich verwenden. Dafür müssen alle diese Cores auf dem selben Rechenknoten liegen
- MPI: Parallelisierung über mehrere Rechenknoten via MPI
Das entsprechende Environment Module „TurboMole/6.5“ erkennt automatisch, in Abhängigkeit von den im Jobskript angeforderten Ressourcen, welche Version genutzt werden kann und setzt die Pfade entsprechend:
- Wenn nur ein Core angefordert wurde, wird die sequentielle Version genutzt
- Bei mehr als einem Core, wird die OpenMP-Version genutzt
- Existiert „mpiprocs=“ im angeforderten Chunk, wird die MPI-Version genutzt
Shared Memory / OMP
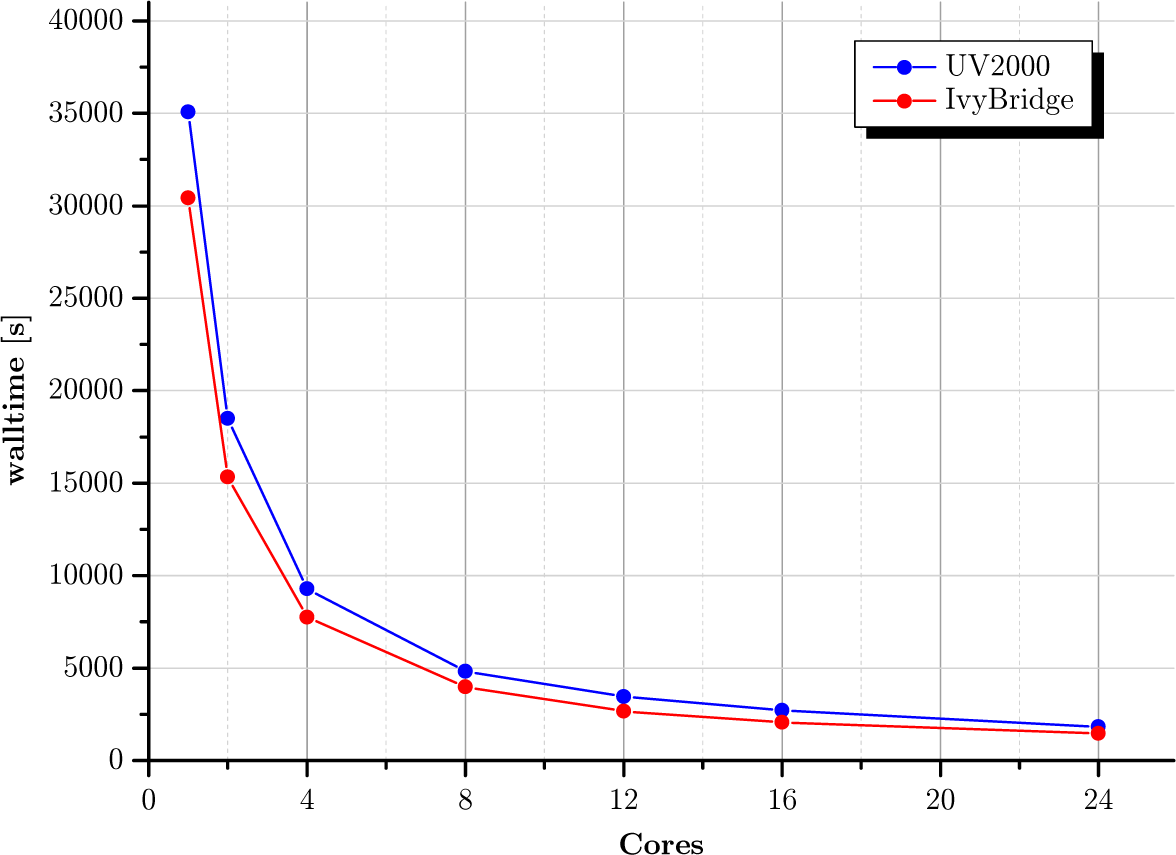
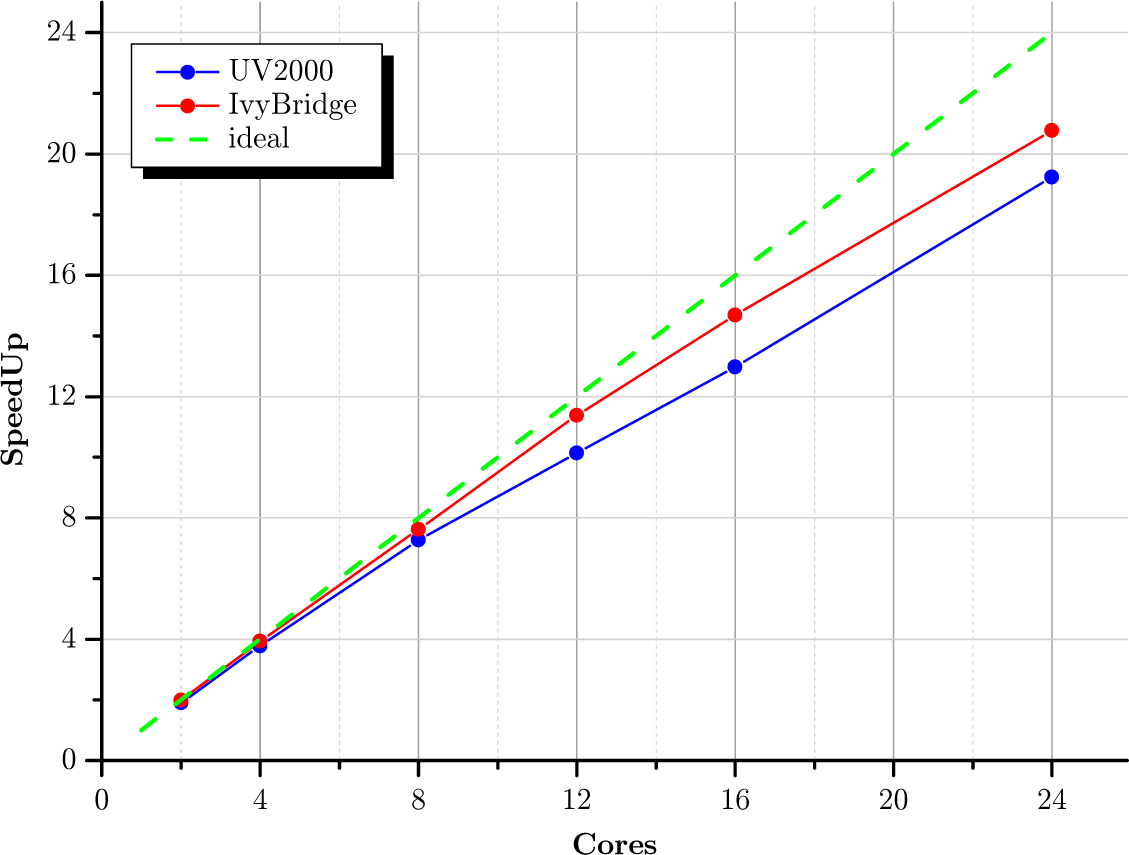
Im Gegensatz zu Gaussian skaliert die OpenMP-Variante von TurboMole exzellent mit der Anzahl der verwendeten Cores. Im Folgenden sind die reine Laufzeit (Walltime) und die erreichte Beschleunigung (SpeedUp) einer DFT-Rechnung (mit dscf_omp) mit steigender Anzahl an Cores für die SGI UV2000 (Sandybridge, 2,4 GHz) und für die Bull Blades (Ivybridge, 2,7 GHz) dargestellt. Die leicht bessere Performance der Ivybridge-basierten Rechenknoten ist allein der höheren Taktrate geschuldet. Dafür stehen auf der UV2000 mit 8 TByte RAM und bis zu 256 Cores gleichzeitig erheblich mehr Ressourcen für Shared-Memory-Rechnungen zur Verfügung.
Rechnung: Guanin-Cytosin (29 Kerne) in der Watson-Creek-Geometrie, DFT, B3LYP, aug-cc-pVTZ an allen Kernen (1104 Atomorbitale, 356 Elektronen).
Job Script Template (Shared Memory / OMP)
#!/bin/bash #PBS -l select=1:ncpus=###CORES###:mem=32gb #PBS -l walltime=999:00:00 #PBS -r n #PBS -N ###JOB NAME### #PBS -A ###myJAM PROJECT### export LOGFILE=$PBS_O_WORKDIR/$PBS_JOBNAME"."$PBS_JOBID".log" #make unique scratch directory on GPFS filesystem SCRATCHDIR=/gpfs/scratch/$USER/$PBS_JOBID mkdir -p "$SCRATCHDIR" cd $PBS_O_WORKDIR echo "$PBS_JOBID ($PBS_JOBNAME) @ `hostname` at `date` in "$RUNDIR" START" > $LOGFILE echo "`date +"%d.%m.%Y-%T"`" >> $LOGFILE #load TurboMole Environment and set scratch directory module load TurboMole/7.3.1 export TURBOTMPDIR=$SCRATCHDIR #copy input files to scratch directory cp -r $PBS_O_WORKDIR/* $SCRATCHDIR/. cd $SCRATCHDIR rm $PBS_JOBNAME"."$PBS_JOBID".log" #some output for debugging CPUSET=/dev/cpuset/PBSPro/$PBS_JOBID CPUSET_CPUS=`cat $CPUSET/cpus` CPUSET_MEMS=`cat $CPUSET/mems` echo >> $LOGFILE echo "GLOBAL PARAMETERS" >> $LOGFILE echo "---------------------------" >> $LOGFILE echo "Node : "`hostname` >> $LOGFILE echo "RunDir : "$PBS_O_WORKDIR >> $LOGFILE echo "TurboBins : "$TURBOBINS >> $LOGFILE echo "ScratchDir: "$SCRATCHDIR >> $LOGFILE echo "# CPUs : "$NCPUS >> $LOGFILE echo " \`-CPUSet: "$CPUSET_CPUS >> $LOGFILE echo " \`-MEMs : "$CPUSET_MEMS >> $LOGFILE echo "# Threads : "$OMP_NUM_THREADS >> $LOGFILE echo "KMP_AFFINITY = "$KMP_AFFINITY >> $LOGFILE echo >> $LOGFILE echo "STARTING TURBOMOLE..." >> $LOGFILE echo "---------------------------" >> $LOGFILE #INSERT TURBOMOLE CALLS HERE, e.g. # actual -r 2>&1 >> $LOGFILE # dscf_omp 2>&1 >> $LOGFILE # or whatever # copy back results to home directory cp -r "$SCRATCHDIR"/* $PBS_O_WORKDIR/. cd $PBS_O_WORKDIR #print the last known statistics of the job (memory usage, cpu time, etc...) echo >> $LOGFILE qstat -f $PBS_JOBID >> $LOGFILE echo "$PBS_JOBID ($PBS_JOBNAME) @ `hostname` at `date` in "$RUNDIR" END" >> $LOGFILE echo "`date +"%d.%m.%Y-%T"`" >> $LOGFILE
Zur Bedeutung und Verwendung der Environment-Variable KMP_AFFINITY siehe: Thread Affinity Interface (Intel)